نوبل شیمی 2024

فاش کردن اسرار پروتئین ها به وسیله محاسبات و هوش مصنوعی!
شیمیدانان مدتهاست به دنبال راهی برای درک کامل و تسلط بر پروتئین ها بودند. این رویا اکنون در دسترس است چراکه Demis Hassabis و John M. Jumper موفق شدند تا از هوش مصنوعی برای پیشبینی ساختار، تقریباً، تمام پروتئینهای شناخته شده استفاده کنند. چرا که David Baker توانست راهی پیدا کند که ساختار پروتئین ها را شناسایی کرده و نیز پروتئین هایی با ساختارهای نو بسازد.
آشنایی با شیمی حیات
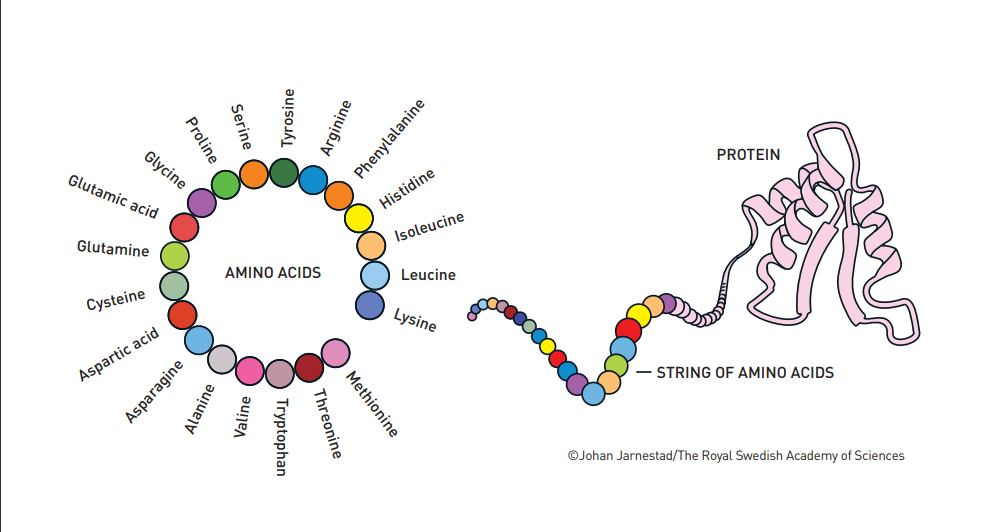
به طور کلی پروتئین ها از 20 نوع اسید آمینه تشکیل شده و به صورت نامحدود با هم ترکیب میشوند. پروتئین ها با استفاده از اطلاعات ذخیره شده در DNA به عنوان یک نقشه، آمینو اسیدها به هم متصل شده تا رشته های طولانی تشکیل دهند.
پس از آن رشته حاوی اسیدهای آمینه، پیچ خورده و تا می شوند و به یک ساختار سه بعدی متمایز و منحصر به فرد تبدیل می شود (شکل 1). این ساختار چیزی است که منجر به عملکرد پروتئین ها میشود. برخی از آنها با قرار گرفتن در کنار سایر اجزای سلولی می توانند ماهیچه ها، شاخ ها یا پرها را ایجاد کنند، در حالی که برخی دیگر ممکن است به هورمون یا آنتی بادی تبدیل شوند. بسیاری از آنها آنزیم هایی را تشکیل می دهند که واکنش های شیمیایی حیات را با دقت شگفت انگیزی هدایت می کنند. پروتئین هایی که روی سطوح سلول ها قرار می گیرند نیز مهم هستند و به عنوان کانال های ارتباطی بین سلول و محیط اطراف آن عمل می کنند.

اهمیت بلوک های شیمیایی تشکیل دهنده ی حیات بر کسی پوشیده نیست. نیمی از جایزه به دمیس حسابیس و جان جامپر تعلق میگیرد که از هوش مصنوعی برای حل موفقیتآمیز مسئلهای که شیمیدانان بیش از 50 سال با آن دست و پنجه نرم میکردندT یعنی پیشبینی ساختار سهبعدی یک پروتئین بوسیله دنبالهای از اسیدهای آمینه، استفاده کردهاند. این به آنها اجازه می دهد تا ساختار تقریباً تمام 200 میلیون پروتئین شناخته شده را پیش بینی کنند. نیمی دیگر از جایزه به دیوید بیکر تعلق می گیرد. او روشهای رایانهای را برای دستیابی به چیزی که بسیاری از مردم معتقد بودند غیرممکن است توسعه داده است؛ یعنی ایجاد پروتئینهایی که از قبل در جهان وجود نداشته و در بسیاری از موارد عملکردهای کاملاً جدیدی دارند.جایزه نوبل شیمی 2024 دو اکتشاف متفاوت را به رسمیت می شناسد، اما همانطور که خواهید دید، آنها ارتباط نزدیکی با هم دارند. برای درک چالش هایی که برندگان امسال برنده شده اند، باید به گذشته تا زمان آغاز بیوشیمی مدرن نگاه کنیم.
اولین عکس ها از پروتئین ها
شیمیدانان از قرن نوزدهم می دانستند که پروتئین ها از مهم ترین اجزای زندگی هستند، اما تا دهه ی 1950 طول کشید تا ابزارهای شیمیایی به قدر کافی دقیق باشند تا محققان با جزئیات بیشتری شروع به کاوش در ساختار پروتئین ها کنند. در پایان این دهه، محققین کمبریج، John Kendrew و Max Perutz، با موفقیت از روشی به نام کریستالوگرافیِ اشعه ایکس برای ارائه اولین مدل های سه بعدی پروتئین ها استفاده کردند و برای قدردانی از این کشف پیشگامانه، جایزه نوبل شیمی در سال 1962 به آنها اعطا شد.
پس از آن محققان در درجه اول از کریستالوگرافیِ اشعه ایکس، که اغلب با زحمت فراوان انجام میشد، برای تولید موفقیت آمیز تصاویر حدود 200،000 پروتئین متفاوت، استفاده کرده اند.
معمای اصلی!
چگونه یک پروتئین ساختار منحصر به فرد خود را پیدا می کند؟
یک دانشمند آمریکایی به نام Christian Anfnsen، موفق شد تا یک پروتئین پیچ و تاب خود را باز کرده و سپسآن را به حالت تا خورده برگرداند. نکته جالب این بود که این پروتئین هر بار دقیقاً به همان شکل پیچ و تاب میخوردند. در نهایت در سال 1961، او نتیجه گرفت که نحوه ی تشکیل ساختار سه بعدی پروتئین کاملاً به اسیدهای آمینه موجود در آن بستگی دارد. این امر باعث شد که وی در سال 1972 جایزه نوبل شیمی را به دست آورد.
با این حال استدلال Anfnsen حاوی یک پارادوکس است، که یکی دیگر از محققان آمریکایی به نام , Cyrus Levinthal، در سال 1969 به آن اشاره کرد. او محاسبه کرد که حتی اگر یک پروتئین فقط از 100 اسید آمینه تشکیل شود، از نظر تئوری می توان برای این پروتئین حداقل 1047 ساختار مختلف سه بعدی را در نظر گرفت. بنابراین اگر زنجیره ای از اسیدهای آمینه به طور تصادفی بچرخد، زمان یافتن ساختار کاربردیِ پروتئین، بیشتر از سن جهان طول می کشد در حالیکه اینکار در یک سلول فقط چند میلی ثانیه طول می کشد. پس چگونه رشته های اسید آمینه در واقع فولد می شوند؟
کشف Anfnsen و پارادوکس Levinthal حاکی از آن است که فرآیند Folding، پروتئین یک فرآیند از پیش تعیین شده است و به طرز حیرت انگیزی تمام اطلاعات مربوط به این فرآیند باید در توالی اسید آمینه وجود داشته باشد.
چالش بزرگ در بیوشیمی
بینش فوق منجر به کشف تعیین کننده دیگری شد. اگر شیمیدانان توالی اسید آمینه موجود در پروتئین را می دانند پس باید بتوانند ساختار سه بعدی پروتئین را پیش بینی کنند. این یک ایده هیجان انگیز بود؛ اگر آنها موفق میشدند، دیگر نیازی به استفاده از کریستالوگرافی با اشعه ایکس نداشته و می توانند در زمان خود را صرفه جویی کنند. آنها همچنین می توانند ساختارهایی را برای تمام پروتئین هایی که کریستالوگرافی اشعه ایکس برای آنها کاربرد ندارد، ایجاد کنند.
این نتیجهگیریهای به یکی از چالش های بزرگ بیوشیمی، یعنی مسئله پیشبینی، تبدیل شد و برای تشویق توسعه سریعتر در این حوزه، در سال 1994 محققان پروژه ای به نام ارزیابی انتقادی پیش بینی ساختار پروتئین (CASP) را آغاز کردند که به یک مسابقه تبدیل شد. در این چالش می بایست ساختارهای پروتئینی را بر اساس توالی اسیدهای آمینه شناخته شده، پیشبینی می شدند. هر دو سال یکبار، محققان از سراسر جهان به توالی هایی از اسیدهای آمینه در پروتئین هایی که ساختار آنها به تازگی تعیین شده بود، دسترسی پیدا می کردند. با این حال، این ساختارها از شرکت کنندگان مخفی نگه داشته شدند.
CASP نظر محققین بسیاری را به خود جلب کرد، اما حل مشکلِ پیش بینی بسیار دشوار بود. مطابقت بین پیشبینیهایی که محققان وارد رقابت کردند و ساختارهای واقعی به سختی بهبود می یافت. اما در سال 2018، زمانی که یک استاد شطرنج، متخصص علوم اعصاب و پیشگام در هوش مصنوعی وارد میدان شد، موفقیت هایی در این پروژه حاصل شد!
ورود استاد بزرگ شطرنج به المپیکِ پروتئین
دمیس حسابیس از چهار سالگی شروع به بازی شطرنج کرد و در سن 13 سالگی به مقام استادی رسید. در نوجوانی او به عنوان یک برنامه نویس و توسعه دهنده موفق بازیها کار خود را آغاز کرد. وی شروع به کاوش در زمینه هوش مصنوعی و علوم اعصاب کرده که منجر به چندین کشف انقلابی شد. او از آنچه در مورد مغز آموخته بود استفاده کرد تا شبکه های عصبی بهتری برای هوش مصنوعی ایجاد کند. در سال 2010 او DeepMind را تأسیس کرد. این شرکت مدلهای AI را برای بازی های محبوب بورد گیم توسعه داد. این شرکت در سال 2014 به Google فروخته شد و دو سال بعد هنگامی که این شرکت به هدف نهایی خود یعنی شکست قهرمان یکی از قدیمی ترین بازی های تخته ای در جهان، یعنی بازی Go رسید، Deepmind مورد توجه جهان قرار گرفت.
با این حال Hassabis معتقد بود که هدف اصلی آنها Go نبود بلکه آن وسیله ای برای توسعه مدل های بهتر هوش مصنوعی بود. پس از این پیروزی، تیم وی آماده مقابله با مشکلات مهم برای دنیای بشریت بود، بنابراین در سال 2018 وی برای رقابت در سیزدهمین دوره از مسابقه CASP ثبت نام کرد.
یک برد غیرمنتظره برای مدل هوش مصنوعی Hassabis
در سالهای گذشته، ساختارهای پروتئینی که محققان برای CASP پیشبینی کرده بودند، در بهترین حالت به دقت 40 درصد دست یافته بود. تیم Hassabis با مدل هوش مصنوعی خود، یعنی AlphaFold، تقریبا به دقت 60 درصد رسید. آن ها پیروز شدند و نتیجه عالی آن بسیاری را غافلگیر کرد. این پیشرفت غیرمنتظره بود، اما راه حل هنوز به اندازه کافی خوب نبود. چرا که برای موفقیت، پیشبینی باید در مقایسه با ساختار هدف، دقت 90 درصدی داشته باشد.
آلفافولد چگونه کار میکند؟
در بخشی از برنامه ی توسعه AlphaFold2، مدل هوش مصنوعی بر روی تمام توالی های اسید آمینه شناخته شده و ساختارهای پروتئینی تعیین شده آموزش دیده است.
- ورود داده ها و جستجو در پایگاه داده
یک توالی اسید آمینه با ساختار ناشناخته به AlphaFold2 وارد میشود و مدل موجود در پایگاههای داده به دنبال توالی های اسید آمینه مشابه و ساختارهای پروتئینی حاصل از آن به جستجو میپردازد.
- آنالیز توالی ها
مدل هوش مصنوعی تمام توالیهای اسید آمینه مشابه، که اغلب از گونههای مختلف هستند را همتراز (Align) کرده و بررسی میکند که کدام قسمتها در طول تکامل محفوظ مانده اند. در مرحله بعد AlphaFold2 بررسی می کند که کدام یک از اسیدهای آمینه می توانند در ساختار پروتئین سه بعدی با یکدیگر تعامل داشته باشند. آمینو اسیدها با هم تکامل می یابند. اگر یکی باردار باشد، دیگری بار مخالف دارد، بنابراین آنها جذب یکدیگر می شوند. اگر یکی با اسید آمینه آبگریز جایگزین شود، دیگری نیز آبگریز می شود.
- تجزیه و تحلیل هوش مصنوعی
با استفاده از یک فرآیند تکراری، AlphaFold2 آنالیز توالی ها و distance map (تخمین میزان نزدیک بودن اسیدهای آمینه در ساختار) را اصلاح می کند. مدل هوش مصنوعی از شبکه های عصبی به نام ترانسفورماتور استفاده می کند که ظرفیت بالایی برای شناسایی عناصر مهم و تمرکز بر روی آن دارند. دادههای مربوط به سایر ساختارهای پروتئینی، اگر در مرحله 1 یافت شوند نیز مورد استفاده قرار میگیرند.
- ساختار فرضی
AlphaFold2 پازلی از تمام اسیدهای آمینه را گرد هم می آورد و مسیرهایی را برای تولید یک ساختار پروتئینی فرضی، آزمایش می کند. انجام دوباره ی مرحله سوم. پس از سه چرخه، AlphaFold2 به یک ساختار خاص می رسد و مدل هوش مصنوعی احتمال مطابقت بخشهای مختلف این ساختار با واقعیت را محاسبه میکند.
Hassabis و تیمش به توسعه AlphaFold ادامه دادند، اما هر چقدر هم که تلاش کردند، الگوریتم آن هرگز کامل نشد. حقیقت تلخ این بود که آنها به بن بست رسیده بودند. تیم خسته بود، اما یک کارمند جدید ایده های قاطعی در مورد چگونگی بهبود مدل هوش مصنوعی داشت. نام آن کارمند جان جامپر بود.
ورود جامپر برای حل چالش بزرگ
شیفتگی جان جامپر به کیهان چیزی بود که او را وادار به مطالعه فیزیک و ریاضی کرد. با این حال در سال 2008، زمانی که در شرکتی شروع به کار کرد که از ابررایانهها برای شبیهسازی پروتئینها و دینامیک آن ها استفاده میکرد، متوجه شد که دانش فیزیک میتواند به حل مشکلات پزشکی کمک کند.
جامپر زمانی که در سال 2011 دکترای خود را در رشته ی فیزیک نظری آغاز کرد، به کار بر روی پروتئین ها ادامه داد. برای صرفه جویی در ظرفیت کامپیوتر، چیزی که در دانشگاه کم بود، شروع به توسعه روش های ساده تر و مبتکرانه تری برای شبیه سازی دینامیک پروتئین کرد. وی پس از مدتی شروع به کار بر روی چالش پیشِ رو کرد. او در سال 2017 به تازگی دکترای خود را به اتمام رسانده بود، هنگامی که شایعاتی مبنی بر اینکه Google DeepMind یه صورت مخفیانه شروع به کار بر روی پیشبینی ساختارهای پروتئینی کرده است، شنید، برایشان درخواست کار فرستاد. تجربه او از شبیهسازی پروتئین به این معنی بود که ایدههای خلاقانهای در مورد چگونگی بهبود AlphaFold داشت، بنابراین پس از مدتی به تیم Hassabis منتقل شد و با هم مشغول به اصلاح مدل موجود شدند.
نتایج خیره کننده با مدل اصلاح شده هوش مصنوعی
نسخه جدید AlphaFold2 با دانش Jumper از پروتئین ها همراه شد. این تیم همچنین شروع به نوآوری حاصل از پیشرفت اخیر در هوش مصنوعی، شبکه های عصبی به نام ترانسفورماتور، کرد. ترانسفورماتورها می توانند الگوهایی را در حجم عظیمی از داده ها به شیوه ای انعطاف پذیرتر از قبل پیدا کرده و به طور موثر تعیین کنند که برای رسیدن به یک هدف خاص باید روی چه چیزی تمرکز کرد.
این تیم AlphaFold2 را با استفاده از اطلاعات گسترده یِ تمام ساختارهای پروتئینی و توالیهای اسید آمینه شناختهشده موجود در پایگاههای اطلاعاتی آموزش دادند و مدل هوش مصنوعی با معماری جدید، شروع به ارائه نتایج خوب و به موقع برای شرکت در چهاردهمین مسابقه CASP کرد.
در سال 2020، زمانی که سازمان دهندگان CASP نتایج را ارزیابی کردند، متوجه شدند که چالش 50 ساله بیوشیمی به پایان رسیده است. در بیشتر موارد، نتایج حاصل از AlphaFold2 تقریباً به خوبی کریستالوگرافی اشعه ایکس است که در نوع خود شگفتانگیز بود. وقتی یکی از بنیانگذاران CASP به نام John Moult مسابقه را در 4 دسامبر 2020 به پایان رساند، از خود پرسید، حالا چه کاری باید انجام شود؟
یک کتاب در مورد سلول باعث تغییر جهت دیوید بیکر شد!
زمانی که دیوید بیکر شروع به تحصیل در دانشگاه هاروارد کرد، فلسفه و علوم اجتماعی را انتخاب کرد. با این حال، در طول دوره ای در زیست شناسی تکاملی، او با اولین ویرایش کتاب درسیِ کلاسیک زیست شناسی مولکولیِ سلول مواجه شد. این باعث شد که او مسیر زندگی خود را تغییر دهد. او شروع به کشف زیست شناسی سلولی کرد و در نهایت مجذوب ساختارهای پروتئینی شد. هنگامی که در سال 1993 به عنوان رهبر گروه در دانشگاه واشنگتن در سیاتل شروع به کار کرد، کار کردن روی چالش بزرگ بیوشیمی را آغاز کرد. او با استفاده از آزمایشهای هوشمندانه شروع به کشف چگونگی تا شدن پروتئینها کرد. این کار به او بینشهایی را داد تا در پایان دهه 1990 شروع به توسعه نرمافزار رایانهای با قابلیت پیشبینی ساختارهای پروتئینی کند؛ این نرم افزار Rosetta نام داشت.
بیکر اولین حضور خود را در مسابقات CASP در سال 1998 با استفاده از Rosetta انجام داد و در مقایسه با سایر شرکت کنندگان، واقعاً خوب عمل کرد. این موفقیت منجر به ایده جدیدی شد؛ اینکه تیم دیوید بیکر می تواند از نرم افزار به صورت معکوس استفاده کند. به جای وارد کردن توالی اسیدهای آمینه در Rosetta و خارج کردن ساختارهای پروتئینی، آنها باید بتوانند ساختار پروتئین مورد نظر وارد کرده و پیشنهاداتی برای توالی اسید آمینه آن بدست آورند. اینکار به آنها اجازه میداد که پروتئین های کاملاً جدیدی ایجاد کنند.
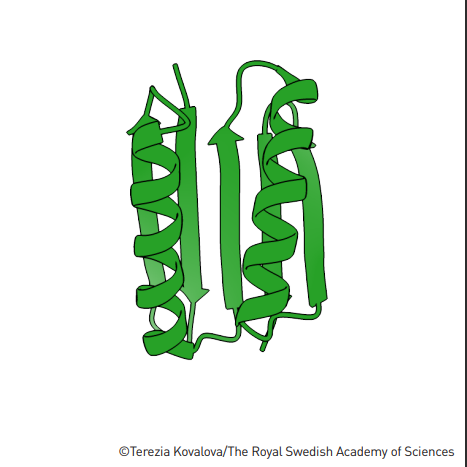
یک پروتئین منحصر به فرد
روشی که در آن پروتئین های کاملا جدید ساخته می شوند طراحی de novo نامیده می شود. گروه تحقیقاتی بیکر، پروتئینی با ساختار کاملاً جدید ترسیم کردند و سپس Rosetta محاسبه و بررسی کرد که کدام نوع از توالی اسید آمینه می تواند پروتئین مورد نظر را ایجاد کند. برای انجام این کار، Rosetta پایگاه داده ای متشکل از تمام ساختارهای پروتئینی شناخته شده را جستجو کرد و به دنبال قطعات کوتاهی از پروتئین هایی بود که شباهت هایی با ساختار مورد نظر داشتند. Rosetta بوسیله ی دانش بنیادی از چشم انداز انرژی پروتئین ها، این قطعات را بهینه کرد و یک توالی اسید آمینه پیشنهاد کرد.
برای بررسی میزان موفقیت نرم افزار، گروه تحقیقاتی بیکر ژن توالی اسید آمینه پیشنهادی را در باکتری هایی که پروتئین مورد نظر را تولید می کردند، القا کرده و سپس ساختار پروتئین را با استفاده از کریستالوگرافی اشعه ایکس تعیین کردند. معلوم شد که Rosetta واقعا می تواند پروتئین هایی با ساختار جدید بسازد. پروتئینی که محققان توسعه دادند، Top7، تقریباً ساختاری داشت که آنها طراحی کرده بودند.
خلاقیت های آزمایشگاه بیکر
Top7 برای محققانی که روی طراحی پروتئین کار میکنند، یک غافلگیری بود. کسانی که قبلاً پروتئینهای de novo را ایجاد کرده بودند، تنها قادر به تقلید از ساختارهای موجود بودند، در حالیکه مشابه ساختار منحصر به فرد Top7 در طبیعت وجود نداشت. همچنین این پروتئین با 93 اسید آمینه خود، بزرگتر از هر چیزی بود که قبلاً با استفاده از طراحی de novo تولید شده بود.
بیکر کشفیات خود را در سال 2003 منتشر کرد. این اولین قدم در زمینه ای بود که فقط می توان آن را به عنوان یک پیشرفت خارق العاده توصیف کرد. تعدادی بسیاری از پروتئین های شگفت انگیز ایجاد شده در آزمایشگاه بیکر را می توان در شکل زیر مشاهده کرد. او همچنین سورس کد Rosetta را منتشر کرد تا جامعه تحقیقاتی جهانی به توسعه نرم افزار ادامه داده و زمینه های کاربردی جدیدی را پیدا کنند.
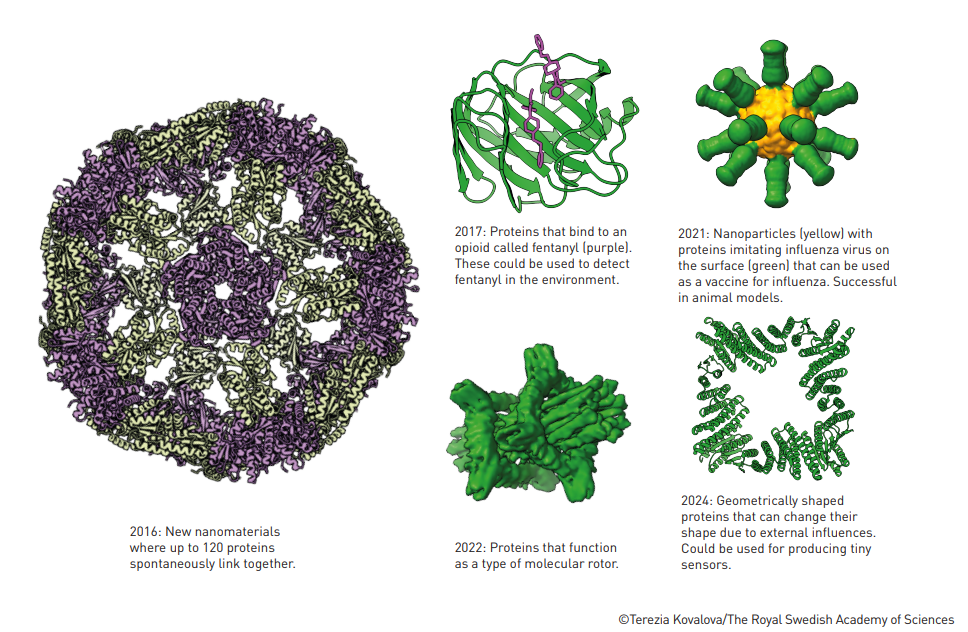
کاری که انجام آن سال ها طول می کشید، اکنون فقط چند دقیقه زمان میبرد!
زمانی که Demis Hassabis و John Jumper تأیید کردند که AlphaFold2 واقعاً کار می کند، ساختار تمام پروتئین های انسانی را ایجاد کردند و سپس ساختار تقریباً تمام 200 میلیون پروتئینی را که محققان تاکنون هنگام نقشه برداری از موجودات زمین کشف کرده اند را نیز پیش بینی کردند.
همچنین Google DeepMind سورس کد AlphaFold2 را در دسترس عموم قرار داده است و همه می توانند به آن دسترسی داشته باشند. تا اکتبر 2024، بیش از دو میلیون نفر از 190 کشور از AlphaFold2 استفاده کردند. تا پیش از این، اغلب سال ها طول می کشید تا ساختار یک پروتئین به دست آید. اکنون می توان آن را در چند دقیقه انجام داد. این مدل هوش مصنوعی کامل نیست، اما میزان درستی و صحت ساختاری را که تولید کرده است تخمین می زند، بنابراین محققان می دانند که این پیش بینی چقدر قابل اعتماد است. شکل زیر چند عدد از نمونه های متعددی را نشان می دهد که چگونه AlphaFold2 به محققان کمک می کند.
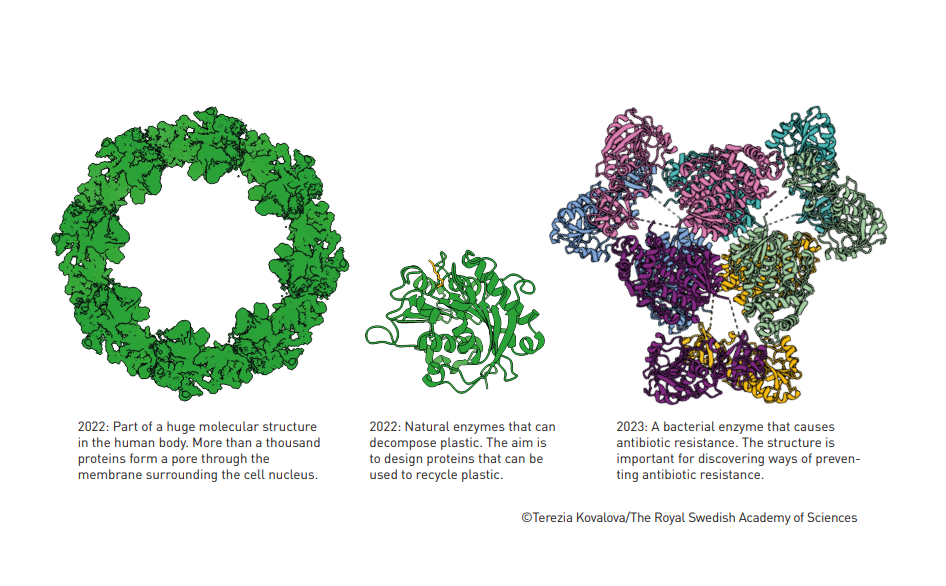
پس از رقابت CASP در سال 2020، زمانی که دیوید بیکر به پتانسیل مدلهای هوش مصنوعی مبتنی بر شبکه ی عصبی ترانسفورماتور پی برد، این مدل را به Rosetta اضافه کرد که طراحی de novo پروتئینها را نیز تسهیل کرده است. در سالهای اخیر تولید پروتئین هایی با ساختارهای باورنکردنی یکی پس از دیگری از آزمایشگاه بیکر انجام شده است.
تطبیقپذیری شگفتانگیز پروتئینها بهعنوان ابزارهای شیمیایی به صورت گستردهای در زندگی ما منعکس شده است. اینکه اکنون میتوانیم به راحتی ساختار این مولکول های کوچک را تجسم کنیم، باعث شگفتی است. چرا که به ما امکان می دهد تا نحوه عملکرد زندگی را بهتر درک کنیم، از جمله اینکه چرا برخی بیماری ها ایجاد می شوند، مقاومت آنتی بیوتیکی چگونه ایجاد می شود یا چرا برخی از میکروب ها می توانند پلاستیک را تجزیه کنند.
نویسنده: زهرا سمیعی

